Dinámica Química Molecular
La simulación a través de la dinámica molecular consiste en una técnica para calcular las propiedades de equilibrio y de transporte de un sistema clásico de muchos cuerpos.
Dado un sistema de N-cuerpos que no posee una solución analítica, la única salida es la solución numérica.
El fundamento para la simulación de dinámica molecular es el conocimiento de la ecuación del movimiento para el sistema considerado. El algoritmo de un programa de dinámica molecular, consiste de la solución numérica de estas ecuaciones de movimiento, otorgando una trayectoria (coordenadas y momentos conjugados en función del tiempo) del sistema en estudio.
Eligiéndose el paso de integración, la resolución temporal y la extensión de la trayectoria pueden ser adaptadas a los ejes de relajación temporal para los procesos dinámicos. A partir de la trayectoria, propiedades de equilibrio y grandezas dinámicas pueden ser calculadas en un código para dinámica molecular.
Una cuestión importante es la relación entre las propiedades de la materia – sea en estado sólido, líquido o gaseoso – y la interacción entre los átomos o moléculas que constituyen este material.
Al contrario de intentar deducir el comportamiento microscópico directamente del experimento, el método de dinámica molecular intenta reproducir el comportamiento utilizando un sistema modelo.
Los métodos de dinámica molecular están basados en los siguientes principios:
* Núcleos y electrones son tratados como partículas tipo átomos
* Estas partículas tipo átomos son esféricas (los rayos son obtenidos de medidas o teóricamente) y tienen una carga líquida (obtenida de la teoría).
* Las partículas interactúan como “resortes” y las interacciones son representadas por potenciales clásicos
* Estas interacciones deben ser predefinidas para conjuntos de átomos específicos
* Las interacciones determinan la distribución espacial de las partículas y sus energías.
El modelo de la dinámica química molecular considera los átomos como esferas y los enlaces como resortes. La matemática de deformación de resortes puede ser utilizada para describir la habilidad de los enlaces en “estirarse” “torcerse” y “voltearse”
El objetivo de la dinámica química molecular es predecir la energía asociada a una dada conformación de una molécula. Con todo, las energías de la dinámica molecular no tienen significado de cantidades absolutas.
Solamente diferencias de energía entre dos o más conformaciones tienen significado físico.
La ecuación para la energía total de un dado sistema en estudio (potencias de interacción) conjuntamente con los datos (parámetros) necesarios para describir el comportamiento de diferentes tipos de átomos y enlaces, es llamado “campo de fuerza”
Modelado de un sistema físico
El principal ingrediente de una simulación es un modelo físico. Para una simulación de dinámica molecular, esto significa elegir un potencial o campo de fuerza: una función V(r1, … rN) de las posiciones de los núcleos, representando la energía potencial del sistema cuando los átomos son acomodados en una configuración específica.
Las fuerzas son entonces deducidas como el gradiente del potencial respecto al movimiento atómico, esto es:
![]()
La elección más sencilla para V es escribirlo como una suma de interacciones de pares.
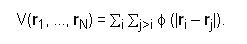
El índice de la segunda sumatoria j > i, nos indica considerar cada par apenas una vez.
La elección del campo de fuerza depende, en gran parte, del sistema a ser estudiado y de las propiedades del mismo que serán investigadas. Más allá una descripción refinada de las vibraciones atómicas sea relevante, las conformaciones de las macromoléculas dependen más de las torciones, de las repulsiones, atracciones de van der waals y de las interacciones electrostáticas.
Optimización de la Geometría Molecular
Calculándose la energía para diversos valores de coordenadas de un dado sistema molecular dependiendo del campo de fuerza al cual está sometido, se puede explorar la superficie de energía potencial para el mismo.
Dependiendo de las dimensiones y características del sistema, la superficie presentará un gran número de mínimos locales de energía que corresponden a puntos en el espacio de configuraciones donde todas las fuerzas sobre los átomos del sistema son balanceadas.
Debido al elevado número de grados de libertad de las macromoléculas biológicas, una exploración completa de la superficie multidimensional de energía es prácticamente imposible.
Una forma de explorar tal superficie se da por la minimización de la energía potencial molecular.
La optimización de la geometría es una técnica que busca encontrar un conjunto de coordenadas que minimizan la energía potencial del sistema de interés. El procedimiento básico consiste en caminar sobre la superficie potencial en la dirección en que la energía decrece de manera que el sistema es llevado a un mínimo de energía local próximo.
Generalmente la configuración final, luego este proceso, no difiere mucho de la inicial.
La minimización de la energía hace uso solamente de una pequeña parte del espacio de configuraciones. Sin embargo, por los ajustes en las posiciones atómicas, se relajan las distorsiones en los enlaces químicos, en los ángulos entre enlaces y en los contactos de van der Waals.
La minimización de energía o atomización de la geometría es un proceso interactivo que en coordenadas cartesianas, puede ser representado por:
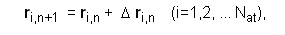
En la cual n es el número de iteraciones y Δ r i , n es el enésimo incremento en las coordenadas del átomo i.
Existen diferentes maneras de determinar el módulo y la dirección del paso (Δ r i , n), los métodos más comunes usan la primer derivada de la función energía potencial, métodos divergentes, en tanto, los métodos más sofisticados usan también la segunda derivada para mejorar la tasa de convergencia.
